PRÁCTICA 14 PCR
INTRODUCCION:
Para la amplificación artificial de ADN o PCR se practicarán protocolos convencionales y la evaluación de los productos por métodos electroforéticos. Con el objetivo de buscar la mayor racionalización en las actividades que se desarrollaran se explican los pasos de las metodologías que se utilizarán.
Para el reporte de práctica se recomienda investigar el marco de referencia teórico de la sesión, así como la introducción para el reporte, por parte del estudiante
Generalidades del PCR
La reacción en cadena de la polimerasa tiene tres pasos que se repiten cíclicamente:
1) desnaturalización de los ácidos nucleicos,
2) alineación de un cebador iniciador (primer), y
3) polimerización, por efecto de una polimerasa termo resistente, en un medio rico de nucleótidos libres (dNTPs).
En un ciclo, a partir de una molécula se obtienen dos, en el segundo ciclo de dos se obtienen cuatro y siguiendo una progresión geométrica en 30 ciclos se tendrían 1, 073, 741, 755 copias. Para una eficiente reacción del PCR, se busca controlar numerosas variables, de las cuales las principales son:
1) Muestra de los ácidos nucleicos,
2) Condiciones químicas de la reacción y
3) condiciones físicas
Muestra de los Ácidos nucleicos
Generalmente los problemas en los resultados del PCR, están relacionados con la cantidad y calidad de ácidos nucleicos. En cuanto a la cantidad, regularmente se sugiere utilizar de 15 a 50 ng, no obstante, para la amplificación de fragmentos de ADN mitocondrial, a partir de muestras de ADN total, se amplía el rango de 15 a 200 ng, prefiriéndose los valores altos. Fuera de estos rangos es necesario utilizar técnicas complementarias o el uso de aditivos en la mezcla de reacción, lo que puede elevar sustancialmente los costos.
En cuanto a la calidad, es preferible utilizar productos del tipo C1 (una banda clara y nítida, sin barrido, ver en interpretación de la electroforesis), aunque en muestras de ADN con barrido ligero (C2 y C3), se pueden obtener resultados adecuados. En muestras degradadas, generalmente se requiere utilizar técnicas complementarias
Condiciones químicas de la reacción
En la mezcla de reacción se utilizan diversos componentes, además del ADN que sirve de molde. Los componentes de una reacción típica, incluyen un cebador hacia adelante (forward o F), uno hacia atrás (reverse o R), mezcla de los dNTPs (deoxinucleotidos), polimerasa termo resistente (Taq polimerasa), iones magnesio (en forma de cloruro de Magnesio), amortiguador y agua destilada, des ionizada, ultra purificada y esterilizada (H2O ddue). Adicionalmente, dependiendo del tipo de termociclador podría utilizarse aceite mineral. La concentración de los cebadores F y R, ha sido ampliamente estandarizada, recomendándose utilizar de 0.5 a 1 µl de una solución con concentración de 20 µM, para una reacción de volumen total de 25 µL. La solución madre (“stock”), se prepara en una concentración de 50 a 100 µM, para garantizar una mejor conservación de los cebadores.
El diseño de los cebadores es básico para entender los resultados de una reacción de PCR. Es recomendable es uso de cebadores específicos, que tengan aproximadamente la misma proporción de C-G con respecto a A-T, evitando la formación de pasadores (dobleces del cebador durante la reacción) o la formación de dímeros simples o pareados (unión de bases complementarias) por unión entre el mismo cebador o entre los cebadores.
Los dNTPs se agregan en exceso para cubrir adecuadamente las demandas de la reacción. En una reacción de 25 µL, es común utilizar 2 µL de una concentración de 2.5mM. Agregar una mayor cantidad implica un desperdicio de los nucleótidos, además de que pueden interferir en la reacción.
La primera polimerasa termo resistente utilizada fue a Taq polimerasa, de la cual se utilizan de 0.25 a 1 unidad por reacción, no obstante, en la actualidad existen al menos 10 polimerasas de uso en las reacciones de PCR, de las cuales las más comerciales son las obtenidas de Pyrococcus furiosus (Pfu), Thermococcus litoralis (Vent o Tli). Generalmente de la solución madre, se utilizan de 0.5 hasta 2 µL por reacción.
El magnesio es requerido para una adecuada función de la polimerasa y se recomienda una concentración de 1.65 mM por reacción. En casos especiales, se puede utilizar hasta 4mM, aunque con el riesgo de disminuir la especificidad en la acción de la polimerasa. La preparación de la solución madre de 25 o 50 mM, es laboriosa y requiere de filtrado y esterilización de los reactivos, las que pueden ser muy difíciles de lograr en un laboratorio general, por lo que se recomienda adquirirla de proveedores especializados.
El pH y el balance iónico, se logra con el uso de un amortiguador termo resistente, con tris-HCl (pH8.4) y cloruro de potasio.
Cabe aclarar que en la reacción debido a los cambios de temperatura el pH variara desde aproximadamente 7 hasta 10. La preparación es sumamente complicada, por lo que se sugiere adquirirlas de una compañía especializada. Finalmente, para el agua, es posible obtenerla de un ultrapurificador y esterilizarla, manteniéndola en el área de trabajo de PCR.
Para lograr una mayor eficiencia en la reacción es posible agregar diversos aditivos, como formamida, DMSO o Tween 20 (se recomienda revisar la página de internet: http://www.staff.uni-mainz.de/lieb/additiva.html), los cuales son utilizados generalmente de forma empírica
Comercialmente se encuentran disponibles juegos de mezclas para PCR, como PCR-super mixer, en los cuales incluyen total o parcialmente los reactivos requeridos, de tal forma que solo se requiere agregar la muestra a amplificar. Lamentablemente en estos juegos, la polimerasa puede perder actividad después de unas semanas o meses y típicamente recomiendan su uso antes de que se cumplan los seis meses de preparación. En los casos que se exceda el tiempo de conservación, suele ser requerido suplementar la mezcla con polimerasa fresca.
En síntesis, la mezcla de reacción típica incluye los siguientes componentes y concentraciones:
Reactivo |
Concentración final |
dNTP"S |
0.2 mM (rango de 0.05 a .25 mM) |
Iniciador F |
0.4 mM (rango de 0.2 a 1 mM) |
Iniciador R |
0.4 mM (rango de 0.2 a 1 mM) |
Taq-Polimerasa |
0.2 U (rango de 0.2 a 1 U) |
Cloruro de magnesio (MgCl2) |
1.65 mM (rango 1.5 a 4 mM) |
ADN (molde o templete) |
15 a 200 ng |
Amortiguador (Tris-HCl 10 a 220 mM pH 8.4- y 50 a 55 mM de KCl), solución madre a 10 X |
1.1 X |
Agua (H2O ddue) |
Volumen necesario para la reacción |
En los primeros termocicladores no se contaban con mecanismos para mantener la temperatura homogénea en todo el interior de los tubos de reacción, por lo que se agrega de uno a dos volúmenes de aceite mineral, previamente esterilizado y conservado en el área de trabajo de PCR.
Condiciones físicas
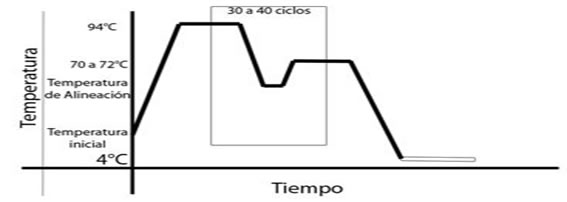
Un ciclo regular requiere de cuatro etapas: 1) desnaturalización inicial, 2) ciclos de desnaturalización, alineación y polimerización, 3) polimerización final y 4) conservación. La desnaturalización inicial, se puede lograr eficientemente con 94 °C durante 5 minutos. Si existe la sospecha de que las muestras no están completamente purificadas (sospecha de proteínas), se puede elevar la temperatura inicial hasta los 96 °C durante 2 minutos y después a 94 °C por los 3 minutos restantes.
Para algunos PCR, como el de RAPDs, puede ser recomendable omitir la desnaturalización inicial. La polimerasa puede perder efectividad durante la desnaturalización inicial, por lo que algunos fabricantes recomiendan agregarla después de esta desnaturalización inicial.
Los ciclos, comprenden una desnaturalización inicial de 94 °C por 30 segundos, la alineación a la temperatura de alineación promedio o de fusión (TM por sus siglas en inglés) por 30 segundos y la polimerización a 72 °C por 1 minuto por cada mil pares de bases.
La temperatura de alineación promedio, se toma a partir de los datos del fabricante de los cebadores o se calcula con la siguiente ecuación empírica:
Calculo de TM (método de G-C)
Por ejemplo, para la secuencia: ttc ccc ggt ctt gta aac c
N= 19 # GC= 10 %GC= 52%
Tm= 2°C * (A+T) + 4°C * (G+C) = 2 * (9) + 4 (10) = 58°C corrección 58-5= 53°C
Nota: Se define TM (Temperatura de fusión) como la temperatura a la cual el 50 % de los ácidos nucleicos se mantiene disociado o desnaturalizado
El número de ciclos está limitado por la eficiencia de la polimerasa, estableciéndose un rango de entre 25 a 40 ciclos. Una mayor cantidad de ciclos puede producir bandas fantasmas u otras aberraciones en los productos del PCR. Las variantes en los ciclos son diversas de acuerdo a las condiciones obtenidas en la estandarización. Las principales variaciones son:
Incrementar en uno a dos segundos por ciclo el tiempo de la polimerización, cuando se obtienen barridos o con baja eficiencia.
Variar la pendiente de cambio entre temperaturas, principalmente entre la alineación y polimerización, cuando hay baja eficiencia en la reacción y en la electroforesis se observan los oligos.Realizar 5 a 6 ciclos iniciales, con 1 a 4 grados arriba de la temperatura de alineación, cuando la reacción es sumamente específica para un fragmento y las falsas alineaciones generan resultados erróneos.
Realizar de 5 a 6 ciclos iniciales, con 1 a 4 grados abajo de la temperatura de alineación, cuando se obtiene baja eficiencia.La polimerización final se realiza a 72 *C por 10 minutos, posteriormente se programa al termociclador en un tiempo de espera a 4 *C y se conservan los productos en refrigerador.
El aceite mineral generalmente no produce problemas en la manipulación de los productos del PCR, no obstante, puede ser necesario eliminarlo. El método más eficiente es utilizar el micropipeteador, otra alternativa es introducir el tubo a -70 °C y una vez solidificada la muestra, se puede retirar fácilmente el aceite por decantación, o deposita el contenido del tubo en un pedazo de parafilm de donde se recoge posteriormente el producto de PCR, los dos últimos métodos involucran un alto riesgo de contaminación.
Otros cuidados
Considerando la gran sensibilidad de la reacción de PCR, se deben extremar los cuidados en:
Proteger de cualquier fuente de contaminación, tanto a los materiales como a los reactivos.
Trabajar con guantes limpios, la protección pertinente y en una campana de flujo laminar tipo I o II.
Hacer reproducibles todas las variables posibles alrededor de los experimentos.
En la extracción de ADN, se debe eliminar los otros compuestos orgánicos presentes en la muestra (residuos de fluidos orgánicos), así como los residuos de los reactivos utilizados en la reacción (SDS, fenol, cloroformo, alcohol y el exceso de sales).
En un estudio, mantener el mismo paquete de materiales y reactivos, ya que el cambio de uno de ellos puede requerir una nueva estandarización.
De las condiciones químicas, la reacción debe contener al menos lo siguiente:
Reactivo |
Concentración esperada |
volumen requerido |
ADN molde |
15 a 50 nanogramos |
|
Cebador RAPD |
0.1 a 1 micromolar |
|
dNTP |
200 micromolar de cada dNTP (dGTP, dCTP, dATP, dTTP) |
|
Taq polimerasa |
0.2 a 1 unidades |
|
Cloruro de Magnesio |
1.5 a 3 molar |
|
Amortiguador |
1.1 X |
|
Agua ddue |
|
|
Aditivos |
variable |
|
Total |
|
25 microlitros |
En un tubo termo transparente se agregan los reactivos necesarios. En el caso de la práctica que realizaremos deberá llevar a un volumen final de 25 microlitros. Posteriormente se homogeneizan los reactivos, se precipitan por centrifugación y en caso de requerirse, se agregan 50 microlitros de aceite mineral para evitar la evaporación durante el experimento. Finalmente, los tubos, debidamente etiquetados se colocan en el termociclador (aparato para controlar las condiciones físicas de la reacción).
Competencia: Valorar diferentes metodologías de la tecnología de ADN a nivel celular para ejemplificar su aplicación, con actitud profesional
Materiales y Equipos
Micropipetas automáticas
¿Qué debe llevar al laboratorio?
ADN
Metodología.
PCR específico
Para los experimentos antes de iniciar la preparación de mezclas, se sugiere determinar el número de muestras que serán analizadas, el volumen de cada reacción individual y preparar una mezcla madre con el total del cebador F, cebador R, dNTPs, magnesio, agua y Taq polimerasa. De la mezcla maestra, son separadas las alícuotas individuales a las que se les agrega la muestra a amplificar.
En el ejercicio se utilizarán cebadores universales para la amplificación de un fragmento especifico de la región control del ADN mitocondrial, que amplifica la región entre el citocromo b y la terminación del D loop 5-CTTGAAAAACCACCGTTGTTA-30) y (50-GTGTTATGCTTTAGTTAAGC-30). De 2354 pares de bases.
Prepare la campana de flujo laminar, encendiendo el aire, limpiando el área de trabajo con alcohol y en su caso se enciende la lámpara UV, y se deja funcionar al menos de 15 a 30 minutos. Recuerde que los rayos UV pueden producir daños en los humanos, por lo que no se olvide de apagar la luz ultravioleta, antes de empezar a trabajar.
Para efectuar el ejercicio amplificación se utilizará un cebador universal
En cuanto a las condiciones físicas, consideramos la desnaturalización, alineación y polimerización en treinta ciclos.
Las condiciones para la amplificación son las siguientes.
Teórico Usado en practica
Desnaturalización inicial 95ºC por 2´45"- Ciclos 30
Desnaturalización 94ºC por 30"
Alineación 50ºC por 30"
Polimerización 72ºC por 30"
- Ciclos 30
- Extensión final 72ºC por 10 minutos
Conservación 16ºC Indefinido
De las condiciones químicas, la reacción se realizará con un paquete comercial, por lo que se debe agregar solo lo siguiente:
Reactivo |
Concentración esperada |
volumen requerido |
ADN molde |
15 a 50 nanogramos |
1 microlitro |
Cebador 1 (forward) |
0.1 a 0.5 micromolar |
1 microlitro |
Cebador 2 (reverse) |
0.1 a 0.5 micromolar |
1 microlitro |
Reactivo comercial |
|
|
Total |
|
25 microlitros |
En un tubo termo transparente se agregan los reactivos necesarios. En el caso de la práctica que realizaremos deberá llevar a un volumen final de 25 microlitros. Posteriormente se homogeneizan los reactivos, se precipitan por centrifugación y en caso de requerirse, se agregan 50 microlitros de aceite mineral para evitar la evaporación durante el experimento. Finalmente, los tubos, debidamente etiquetados se colocan en el termociclador (aparato para controlar las condiciones físicas de la reacción).
Recomendaciones para plasmar los resultados y su análisis
Describa el trabajo realizado en la campana, analice lo realizado (¿qué reactivos utilizo?, ¿porque?).

